Seminar „Genetischer Code und Genomgrammatik“ (SS2011)
Einer der effizientesten bekannten Informationsspeicher ist die DNA. Inhalt dieses Seminars ist es, die biochemischen und biophysikalischen Grundlagen zu beleuchten und die Prinzipien der Verarbeitung genetischer Information („Replikation“ und „Genexpression“) als Basis allen Lebens zu verstehen. Daneben sollen Gen- und Genomsequenzen als spezielle Strings betrachtet und ein Überblick über statistische Auswertungsverfahren von Gensequenzen als wichtiges Teilgebiet der Bioinformatik erarbeitet werden.
Je nach Interessenlage und Kenntnisstand der Teilnehmerinnen und Teilnehmer können auch Antworten auf Fragen gesucht werden wie: Wieso basiert die genetische Information auf einem 2-Bit-Alphabet? Gibt es eine Grammatik des Lebens? Sind Biocomputer die besseren Rechner?
Zeitplan
| Datum | Uhrzeit | Thema | Vortragende(r) |
|---|---|---|---|
| 1.4.2011 | 11.15 | Vorbesprechung | — |
| bis 15.4. | — | Vereinbarung der Gliederungen | — |
| 26.5. | 09.00 |
Chemische Grundlagen des Lebens
|
Christina Funke |
| 26.5. | 09.45 | Statistische Betrachtungen von menschlichen Genomsequenzen | Cem Kiyak |
| 26.5. | 10.30 |
Grammatik der Genome
|
Andreas de Vries |
| bis 6.6. | — | Einreichung der Hausarbeit | — |
Hintergrundinformationen
-
Download der menschlichen Genomsequenzen per FTP:
ftp://ftp.ncbi.nih.gov/genomes/H_sapiens/
(User: anonymous, kein Passwort). Sinnvoll sind die Dateien mit der Endung
.faim FASTA-Format, gezippt also mit der Endung.fa.gz. Die größten menschlichen Chromosomen sind Nr. 1 und 2 mit ungezippt jeweils etwa 220 MB und 230 MB, die kleinsten Nr. 21 und 22 mit etwa 34 MB und das Y-Chromosom mit 25 MB.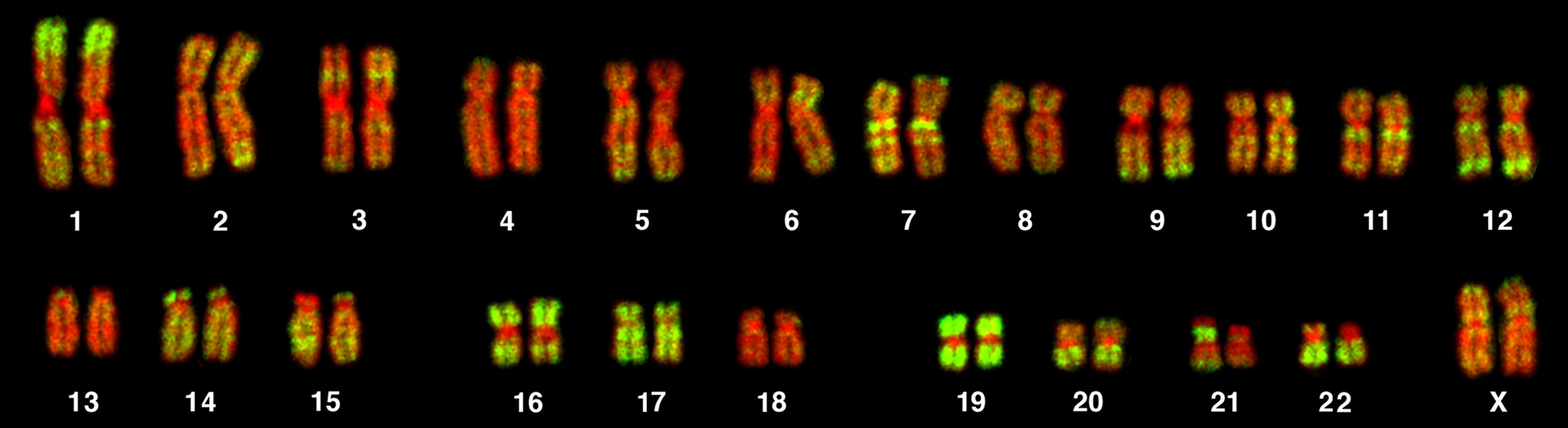
Auf Metaphasechromosomen aus einem menschlichen weiblichen Lymphocyten wurden durch Fluoreszenz in situ Hybridisierung die Alu-Sequenzen markiert (grün). Diese Sequenzen sind besonders in genreichen Chromosomenabschnitten häufig. DNA ist rot eingefärbt.
(Quelle: http://upload.wikimedia.org/wikipedia/commons/2/27/PLoSBiol3.5.Fig7ChromosomesAluFish.jpg) -
Gerne stelle ich das Programm
Utilsin dem Paketorg.mathIT.genesmeiner Java-Bibliothek http://www.math-it.org/java/ zur Verfügung, mit der einfache statistische Auswertungen von FASTA-Dateien durchgeführt werden können, z.B. eine Liste aller Worte der Länge k, die nicht vorkommen, oder eine Häufigkeitsverteilung der verschiedenen Buchstabenkombinationen. Manche der Programme sind momentan noch eine Baustelle und somit nicht überall leicht nachvollziehbar ... - Für die an formalen Sprachen Interessierten: Ich habe versucht, das für das Seminar Wichtigste möglichst knapp zusammenzutragen, siehe languages.pdf. Auch hier gilt jedoch: Vorsicht Baustelle!

